数字化和综合病理学在易位检测中的作用:叙述性回顾
简介
数字病理学由不同的技术组成,这些技术能够将“经典”工作流程转变为全自动和可跟踪的路径[1],最终使用全玻片成像(WSI)进行初步诊断、会诊或多学科讨论[2-4]。尽管它可以代表一种宝贵的信息来源和具有高回报的长期投资,但数字病理学的推进仍旧缓慢,导致一些作者开始推测缓慢推进的可能原因[5]。为了解决这些困惑并为这一过渡提供便利的逐步指导,一些国际协会发布了有关逐步实施数字病理学的详细建议和指南[6,7]。这些可以代表进一步部署病理学“第三次革命”的基础[8],其代表是人工智能(AI)工具在临床和研究活动中的应用[9]。根据不同亚专业(例如放射学、分子生物学和免疫肿瘤学)之间的必要整合,最终综合表征癌症[10],数字病理学可以发挥新的关键作用。因此,本综述将重点关注数字和计算病理学在检测和预测易位方面的进展,这可能与接受分子治疗的候选患者的特定靶点相关。我们根据Narrative Review报告清单(可在https://pcm.amegroups.com/article/view/10.21037/pcm-21-56/rc获得)呈现以下文章。
方法
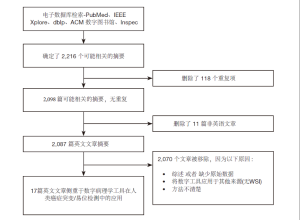
使用以下术语在不同的数据库(PubMed、IEEE Xplore、dblp、ACM数字图书馆、Inspec)上进行系统的文献检索:“digital pathology AND translocations”“digital pathology AND mutations”“artificial intelligence AND translocations”“deep learning AND translocations”“deep learning AND mutations”“digital pathology AND lung cancer”,从2006年1月1日到2021年10月31日,为期15年(表1)。该研究产生了2,216个结果,其中删除了118个重复项和11篇非英语文章(图1)。对于获得的2 087篇文章,两位作者(CB和SC)对摘要进行了评估,并筛选了专注于从数字病理学工具检测突变/易位的手稿。稿件选择的不一致问题已通过第三作者(VL或FP)的重新评估得到解决。对于这些文章(n=17),完整的手稿已经过修改,并且从参考文献中增加了额外的文章到本综述中。

Full table
数字病理学工作流程:AI实施的基本要求
实施WSI作为载玻片的替代物只是向数字病理学工作流程过渡所需的步骤之一[11]。实际上,正如最近的建议所强调的那样,从加入到报告和存档阶段开始,这代表了应该影响解剖病理学实验室的渐进式修改的最后阶段[7]。在这个方向上最重要的进步之一是引入了完全跟踪和完全自动化的实验室工作流程,并辅以条形码的实施,以在诊断过程中唯一地识别每个病例/患者。通过在总装站、嵌入站和切片站[1]使用专用的条形码阅读器和摄像头进一步增强了这一点,有助于显著降低由于WSI上的材料缺失/代表性不足而导致的错误率。与实验室信息系统(LIS)的完全集成进一步促进了这一点,这是一个基本接口,在病理检查的所有阶段发挥不同的作用,包括样本和测试订单输入、样本处理和跟踪。此外,采用以LIS为中心的方法可以轻松地将WSI集成到每位病理学家的诊断程序中,而独立于实验室中使用的扫描仪和用品供应商。这些是采用AI工具检测人类癌症中的突变/易位的主要和基本要求。
为数字病理学增加分子维度:癌症突变的检测
人工智能工具与数字病理学的应用已经证明在传统组织学诊断中具有互补作用[12-14]。 此外,一种根据原发性肿瘤的苏木精和伊红(H&E)染色切片预测转移侵袭性行为的应用已经被评估[15]。最令人着迷的方面是从表型数据预测特定癌症相关突变的可能性。这并不奇怪,因为肿瘤细胞表型(细胞核和细胞质结构、大小、形状)毕竟是由遗传改变赋予的,因此,AI可以通过观察H&E幻灯片的形态模式直接预测单个基因突变。应该注意的是,成像技术只能识别能够直接影响组织形态的遗传变异,AI算法并不能应用于等位基因特定突变的实际变异频率影响疾病的分类和预后的情况,比如一些血液系统肿瘤[16]。Chen等人利用H&E染色的肝细胞癌(HCC)的WSIs训练的卷积神经网络(CNN)证明了HCC中10个最常见的预后和突变基因中的4个(CTNNB1、FMN2、TP53和ZFX4)是如何通过AI正确识别的[17]。他们的方法在识别正常肝组织和肿瘤方面表现出很高的性能(AUC=0.961;95%CI:0.939~0.981)。此外,在区域/瓦片水平上测试模型的突变预测能力时,10个最显著的基因突变中的5个,包括ARID1A(p=0.036)、CTNNB1(p<0.0001)、FMN2(p= 0.0003)、TP53(p=0.0011)和ZFX4(p=0.0054),在突变组和野生型组之间显示出显著差异,外部验证集的AUC为0.71~0.89。Tsou等人基于H&E切片上选定的ROI,训练了CNN来预测甲状腺乳头状癌(PTC)、BRAF V600E和RAS中最常见的致癌驱动因素。他们的工作取得了良好的结果,有趣的是,该模型对RAS突变的预测性能比对BRAF突变的预测性能更好,BRAF组和RAS组的成功预测率分别为63.6%和90%[18]。事实上,这种方法的未来潜在应用可能会减少通常因需要基因检测而延迟的周转时间,并实施反射测试方法,将分子确认限制于算法强调的那些病例。通过接近疾病的分子评估而不考虑癌症起源部位,以完全不可知论的方式行动是最终目标。对于微卫星不稳定性(MSI)状态,深度学习(DL)已用于不同的恶性肿瘤,如区分胃癌和结直肠癌样本中MSI的预测特征[19],甚至与错配修复蛋白免疫组织化学(IHC)和MSI−PCR相比,基于H&E染色的WSI在预测MSI状态方面显示出了更高的性能(AUC为0.865vs.0.605)[20]。
为数字病理学增加分子维度:在易位检测中的可能作用
使用数字/综合病理学工具研究易位检测的首批领域之一可能以血液肿瘤学为代表,其中不同病例的特征是特定的、有时是特征性的染色体重排。95%的急性早幼粒细胞白血病(APL)病例携带15号染色体和17号染色体之间的染色体平衡易位t(15;17)(q24;q21),导致PML−RARa融合蛋白的形成[21,22]。分子技术需要时间,并且在许多医疗资源有限的国家无法使用。相反,外周涂片是普遍可用的,并且在所有医疗保健环境中都能够容易且快速地获得。由于这些原因,一些研究者研究了DL方法是否可以找到预测分子易位状态的形态学特征,以便从细胞形态学中正确鉴定APL与其他亚型的髓系白血病[23]。基于82例患者的发现队列上提出的模型,在24例患者的独立前瞻性验证队列上测试,当分析仅限于未成熟髓系细胞时,发现队列ROC曲线的AUC值为0.822,验证队列ROC曲线的AUC值为0.739,当考虑全部细胞时,性能较低。有趣的是,对于表型分类,AI集中于非APL白血病细胞的细胞质像素和APL细胞的细胞核像素,提示Wright染色的染色质的分布存在一些差异,在非APL细胞中可能更集中于边缘,而APL细胞中更集中于中心。在弥漫性大B细胞淋巴瘤(DLBCL)中,癌基因MYC的重排的预后价值较差,但当它与BCL2/BCL6重排相关时,定义了高级别淋巴瘤的一个亚群[24]。尽管MYC的IHC已被提议作为替代标记物,但仍缺乏可靠的临界值来降低观察者间的重复性误差并提高该标记物的性能[25]。出于这个原因,Swiderska-Chadaj等人基于91个病例的多中心数据的发现队列和66个WSI的测试队列开发了一个DL模型,用来检测来自DLBCL的WSI中的MYC重排[26]。这种方法的准确度为0.77,敏感性和特异性分别为0.88和0.66。他们进一步使用CD20的IHC来定义肿瘤区域,在更大的队列(245例患者)中进一步验证了他们的发现,内部和外部验证集的敏感性和特异性分别为0.90/0.95和0.52/0.53[27]。与MYC的IHC相比,该算法给出了快速和廉价的预筛选,可以指导MYC重排的基因检测的应用。MYC的IHC显示出较低的灵敏度(IHC为0.86,该算法为0.93)和较低的特异性(IHC为0.36,该算法为0.52)[28]。这种方法最有趣和最令人兴奋的方面之一是,MYC的IHC的“假阴性”结果可以通过AI模型的高灵敏度来弥补,使其能够检测到更多的MYC重排患者。用于检测易位的AI工具的使用范围不仅仅限于血液肿瘤学领域,一些实体瘤已经体验到这种数字/综合病理学技术的好处。例如,肾细胞癌(RCC)的一些特定亚型以特定的染色体易位为特征(例如,Xp11.2导致涉及TFE3蛋白的融合转录物),显示出与最常见的透明细胞组织型难以区分的形态学特征[29,30]。在这种情况下,Cheng等人基于TFE3-RCC和透明细胞RCC(ccRCC)的WSI的比较,应用机器学习模型来识别Xp11.2易位[31]。从两个不同的H&E染色的WSI数据集(总共148个图像)开始,TFE3−RCC—ccRCC比率为1:1(TFE3−RCC诊断经FISH确认),他们发现AUC范围为0.842~0.894,强调了该模型捕捉TFE3−RCC和ccRCC之间细微形态差异的能力。最后,这种数字病理学工具的应用甚至可以用于促进检测某些染色体异常的常规测试(例如,荧光原位杂交FISH)的评估,如在乳腺癌中,在其中将近20%的侵袭性肿瘤中发现HER2基因扩增,并且具有这种改变的患者能够受益于靶向治疗[32]。HER2重排是通过用IHC评估HER2状态和用ISH反射检测可疑的IHC(2+)来确定的。尽管一些肿瘤学会强调需要通过FISH程序(HER2以及ALK、ROS1和NTRK)的自动化来实现标准化[33],但这些切片仍主要通过荧光显微镜进行人工分析。一些作者已经尝试使用全自动FISH程序验证HER2基因状态的标准化检测,在Leica FISH(自动)和Abbott FISH对一组328例浸润性乳腺癌进行修正后,获得了99.4%(κ=0.97)的总体一致性[34]。此外,在FISH数字化玻片上应用进一步的人工智能算法可以提供良好的图片质量,与手动FISH相比,减少了分析时间,并且提供了更为简单的图片存档和远程诊断,使HER2 FISH评估更有效[35]。
为数字病理学增加分子维度:聚焦肺癌及其易位
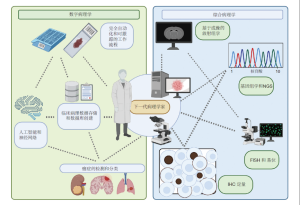
在过去的20年中,对非小细胞肺癌(NSCLC),特别是对腺癌的异质性的重要见解催生了新的研究领域,旨在阐明肿瘤细胞和其周围肿瘤微环境的其他可操作特征。在精准医疗时代,靶向体细胞突变的识别是确定肺源性腺癌患者最佳全身治疗的重要步骤。在这些年里,人们已经做出了不同的努力来破译可以支撑NSCLC特定基因改变的细微形态差异[36,37]。 在这种情况下,创新AI技术的应用代表了一种有希望的信息来源,可以补充“经典”组织病理学,正如这些工具正确检测[38]和分类NSCLC亚型[9]的能力所证明的那样,可以预测患者预后[40]和转移性淋巴结的存在。最近,Coudray等人使用深度CNN正确地将肺肿瘤分类为腺癌和鳞状细胞癌,AUC为0.97(相对于人眼),并通过分析AUC为0.733~0.856的WSI预测了10个突变中的6个(STK11、EGFR、FAT1、SETBP1、KRAS和TP53)[42]。这可以在将来选择性地应用于活检材料不足以进行额外的突变分析的脆弱患者,作为液体活检的可能替代方案。尽管在过去的十年中,人们已经提出了不同的自动化系统来扫描FISH样本并对其进行评分,以评估ALK[43]、ROS1、RET和NTRK畸变[44],从而改变数字化FISH分析的最佳工作流程[35],但通过AI工具从扫描的H&E玻片中预测染色体畸变的尝试仍然非常缺乏。然而,该领域是有希望的,这可以通过在其他领域(如甲状腺癌)检测到其中一些融合的可能性来证明。事实上,从802个甲状腺癌H&E染色的WSI(其中23个属于NTRK融合的肿瘤)的队列开始,提出的预测NTRK状态的CNN模型在来自用于训练的同一队列的图像上显示出大于0.8的AUC[45]。然而,模型性能在外部队列中显著下降。这种方法另一个有希望的前沿表现为整合方式,将基于组织学的人工智能与来自其他亚专业的信息相结合[46-47]。在这种情况下,一些研究小组已经证明了来自计算机断层扫描(CT)[48]或正电子发射断层扫描/计算机断层扫描(PET/CT)[49]的放射组学与临床病理学数据相结合来预测ALK重排存在的能力,再次强调了病理学综合作用的重要性和数字病理学的巨大潜力(图2)。
该方法的局限性和挑战
迄今为止,应用AI工具预测不同类型癌症的突变/易位所获得的结果令人兴奋,但仍然存在一些缺陷,在设计研究预案和更严格的研究时应考虑到这些缺陷。首先,影响这些研究的常见局限之一是所用队列的维度,尤其是验证队列的维度,通常较小且富含最“常见”的癌症组织类型,这可能无法确保涵盖临床实践中可能遇到的整个肿瘤异质性,可能会阻碍对不太常见的组织学类型的研究,从而限制算法在应用于这些罕见肿瘤时的性能。此外,应该始终注意的是,该模型不能完全替代病理学家的检查,其中包括病理学家通常检查的组织多样性和异质性(例如炎症、坏死和血管)。为了使AI算法更具有“分析”性质,其他的挑战应该被注意到,首要的是标准化WSI的污点[50],目前存在不同的方法(例如,度量学习、领域不可知论学习、迁移学习或持续学习),应该在未来的研究中进行相应的调整。此外,人工智能领域的新方法可以帮助更好地理解驱动模型决策的视觉特征,在识别特定的形态特征方面,未来甚至可以通过人眼就可以预测潜在的遗传景观。最后,不同研究之间的分割过程仍然存在很大差异,一些作者执行手动ROI识别,既费时又容易出现人为错误。从这些方面来看,与基于自动分割的研究[17,20]相比,那些使用“手动””方法[18,19]的研究可能会受到一些偏见的影响,因为分析的“监督”特征可能会影响最终结果。
在常规临床实践中转化AI工具
为了能够预测临床上常见的遗传异常,这些研究的结果应当遵循监管机构规定的要求。从这个方面来看,为了能被美国食品药品管理局(FDA)批准为体外诊断工具[51],这种基于AI的设备应当遵循严格的方法学建议,可总结如下:
- 技术性能评估和研究协议(例如,ROI定位研究),以及用于评估设备输出的结果(例如,图像叠加、图像热图等),应明确声明并遵循最严格的证据;
- 训练数据集必须包括代表不同预分析变量的案例,这些变量代表按预期使用时可能遇到的情况(例如,固定类型和时间、组织学幻灯片处理技术、多个站点、患者人口统计数据、具有挑战性的诊断案例,尤其是在处理罕见的遗传变异时);
- 独立验证数据集中的WSI数量必须适合证明设备在检测和定位扫描WSI上的ROI方面的准确性,并且必须包括与临床上设备预期用途相关的子集。
另一个可以显著影响此类AI预测工具性能的关键点,是在研究的训练阶段由算法自动从WSI检索到的瓦片/补丁的大小和数量。最近,将WSI分解为许多较小的可管理的图像块,从而进行高通量采样,这使研究人员能够替换仅能分析小ROI而无法处理大型WSI的旧的AI方法。虽然增加用于训练的图像块大小可能会提高算法性能[52],但这也可能会增加计算时间和成本。因此,独立于实际使用的补丁数量,最重要的步骤是确保训练集中样本/图片多样性的良好表征,因为在单个目标间可能存在大量差异。关于所采用的WSI的尺寸单块和放大倍数,在本综述中分析的研究中情况非常不同,一些处理20倍[17,19,26,42]的扫描载玻片,另一些处理40倍的[31],提取不同大小的区域为0.5 μm/px或0,24 μm/px (MPP):2 000×2 000[31],1 500×1 500[19],512×512[18,27,42],360×360[23]和256×256[17]。然而,所有这些研究通用一个共同的方法,即排除受处理伪影影响的区域或避免使用具有少量信息的补丁(例如,所有>50%的表面被背景覆盖的瓦片,即在RGB颜色空间中值低于220)。这些方法似乎与主要监管机构[51]已批准的几种体外诊断设备数据表中提出的方法一致,这些设备已在临床实践中用于形态学检测WSI中的肿瘤区域。最后,最近的经验强调了执行无注释WSI训练方法的可能性,该方法不需要检测补丁/切片,并专门在肺癌标本上进行测试,这将进一步简化工作流程并减少研究之间的变异性[53]。这些技术方面的逐步清晰和标准化有助于显著增加应用AI工具进行癌症预测的临床试验数量。值得注意的是,正在进行的研究(ClinicalTrials.gov Identifier:NCT04217044)正在评估使用来自常规H&E的AI模型预测神经胶质瘤最常见基因改变(1p/19q 共缺失、MGMT 甲基化、IDH和TERTp突变等)的可能性。这一巨大转变的后续步骤将是在日常实践中使用此类工具,供不熟悉复杂信息学和AI的病理学家和肿瘤学家使用。因此,未来的挑战将是将这些预测算法完全集成到当前使用的自动化工作流程中[3],正如临床级的计算病理学中基于AI的诊断工具所经历的那样[14,54]。
结论
数字病理学正在成为预测不同类型肿瘤基因畸变的宝贵工具,在肺癌领域中具有广阔的应用前景。真正将分子检测范式从手动方法转变为全自动方法的原因在于引入用于IHC和FISH分析的数字扫描系统,而在这种情况下实施人工智能,尽管有希望,但仍需要进一步研究。
Acknowledgments
We would like to thank Dr. Umberto Malapelle and Dr. Pasquale Pisapia from the University of Naples Federico II for their support in the creation of the iconography of this manuscript.
Funding: None.
Footnote
Provenance and Peer Review: This article was commissioned by the Guest Editors (Fabrizio Tabbò, Umberto Malapelle, Maria Lucia Reale, and Angela Listì) for the series “How to Detect and Treat NSCLC Patients With Oncogenic Fusions” published in Precision Cancer Medicine. The article has undergone external peer review.
Reporting Checklist: The authors have completed the Narrative Review reporting checklist. Available at https://pcm.amegroups.com/article/view/10.21037/pcm-21-56/rc
Conflicts of Interest: All authors have completed the ICMJE uniform disclosure form (available at: https://pcm.amegroups.com/article/view/10.21037/pcm-21-56/coif). The series “How to Detect and Treat NSCLC Patients With Oncogenic Fusions” was commissioned by the editorial office without any funding or sponsorship. The authors have no other conflicts of interest to declare.
Ethical Statement: The authors are accountable for all aspects of the work in ensuring that questions related to the accuracy or integrity of any part of the work are appropriately investigated and resolved.
Open Access Statement: This is an Open Access article distributed in accordance with the Creative Commons Attribution-NonCommercial-NoDerivs 4.0 International License (CC BY-NC-ND 4.0), which permits the non-commercial replication and distribution of the article with the strict proviso that no changes or edits are made and the original work is properly cited (including links to both the formal publication through the relevant DOI and the license). See: https://creativecommons.org/licenses/by-nc-nd/4.0/.
References
- L'Imperio V, Gibilisco F, Fraggetta F. What is Essential is (No More) Invisible to the Eyes: The Introduction of BlocDoc in the Digital Pathology Workflow. J Pathol Inform 2021;12:32. [Crossref] [PubMed]
- Fraggetta F, Garozzo S, Zannoni GF, et al. Routine Digital Pathology Workflow: The Catania Experience. J Pathol Inform 2017;8:51. [Crossref] [PubMed]
- Fraggetta F, Caputo A, Guglielmino R, et al. A Survival Guide for the Rapid Transition to a Fully Digital Workflow: The "Caltagirone Example". Diagnostics (Basel) 2021;11:1916. [Crossref] [PubMed]
- L'Imperio V, Brambilla V, Cazzaniga G, et al. Digital pathology for the routine diagnosis of renal diseases: a standard model. J Nephrol 2021;34:681-8. [Crossref] [PubMed]
- Griffin J, Treanor D. Digital pathology in clinical use: where are we now and what is holding us back? Histopathology 2017;70:134-45. [Crossref] [PubMed]
- Evans AJ, Brown RW, Bui MM, et al. Validating Whole Slide Imaging Systems for Diagnostic Purposes in Pathology: Guideline Update From the College of American Pathologists in Collaboration With the American Society for Clinical Pathology and the Association for Pathology Informatics. Arch Pathol Lab Med 2021; [Epub ahead of print].
- Fraggetta F, L'Imperio V, Ameisen D, et al. Best Practice Recommendations for the Implementation of a Digital Pathology Workflow in the Anatomic Pathology Laboratory by the European Society of Digital and Integrative Pathology (ESDIP). Diagnostics (Basel) 2021;11:2167. [Crossref] [PubMed]
- Salto-Tellez M, Maxwell P, Hamilton P. Artificial intelligence-the third revolution in pathology. Histopathology 2019;74:372-6. [Crossref] [PubMed]
- Holzinger A, Malle B, Kieseberg P, et al. Machine Learning and Knowledge Extraction in Digital Pathology Needs an Integrative Approach. Towards Integrative Machine Learning and Knowledge Extraction 2017:13-50. Available online:
10.1007/978-3-319-69775-8_2 10.1007/978-3-319-69775-8_2 - Bera K, Schalper KA, Rimm DL, et al. Artificial intelligence in digital pathology - new tools for diagnosis and precision oncology. Nat Rev Clin Oncol 2019;16:703-15. [Crossref] [PubMed]
- Fraggetta F, Pantanowitz L. Going fully digital: utopia or reality? Pathologica 2018;110:1-2. [PubMed]
- Sudharshan PJ, Petitjean C, Spanhol F, et al. Multiple instance learning for histopathological breast cancer image classification. Expert Syst Appl 2019;117:103-11. [Crossref]
- Xu Y, Zhu JY, Chang EI, et al. Weakly supervised histopathology cancer image segmentation and classification. Med Image Anal 2014;18:591-604. [Crossref] [PubMed]
- Campanella G, Hanna MG, Geneslaw L, et al. Clinical-grade computational pathology using weakly supervised deep learning on whole slide images. Nat Med 2019;25:1301-9. [Crossref] [PubMed]
- Ehteshami Bejnordi B, Veta M, Johannes van Diest P, et al. Diagnostic Assessment of Deep Learning Algorithms for Detection of Lymph Node Metastases in Women With Breast Cancer. JAMA 2017;318:2199-210. [Crossref] [PubMed]
- Sallman DA, Padron E. Integrating mutation variant allele frequency into clinical practice in myeloid malignancies. Hematol Oncol Stem Cell Ther 2016;9:89-95. [Crossref] [PubMed]
- Chen M, Zhang B, Topatana W, et al. Classification and mutation prediction based on histopathology H&E images in liver cancer using deep learning. NPJ Precis Oncol 2020;4:14. [Crossref] [PubMed]
- Tsou P, Wu CJ. Mapping Driver Mutations to Histopathological Subtypes in Papillary Thyroid Carcinoma: Applying a Deep Convolutional Neural Network. J Clin Med 2019;8:1675. [Crossref] [PubMed]
- Kather JN, Pearson AT, Halama N, et al. Deep learning can predict microsatellite instability directly from histology in gastrointestinal cancer. Nat Med 2019;25:1054-6. [Crossref] [PubMed]
- Yamashita R, Long J, Longacre T, et al. Deep learning model for the prediction of microsatellite instability in colorectal cancer: a diagnostic study. Lancet Oncol 2021;22:132-41. [Crossref] [PubMed]
- Wang ZY, Chen Z. Acute promyelocytic leukemia: from highly fatal to highly curable. Blood 2008;111:2505-15. [Crossref] [PubMed]
- Mosleh M, Mehrpouri M, Ghaffari S, et al. Report of a new six-panel flow cytometry marker for early differential diagnosis of APL from HLA-DR negative Non-APL leukemia. Scand J Clin Lab Invest 2020;80:87-92. [Crossref] [PubMed]
- Sidhom JW, Siddarthan IJ, Lai BS, et al. Deep learning for diagnosis of acute promyelocytic leukemia via recognition of genomically imprinted morphologic features. NPJ Precis Oncol 2021;5:38. [Crossref] [PubMed]
- Xie Y, Pittaluga S, Jaffe ES. The histological classification of diffuse large B-cell lymphomas. Semin Hematol 2015;52:57-66. [Crossref] [PubMed]
- Ambrosio MR, Lazzi S, Bello GL, et al. MYC protein expression scoring and its impact on the prognosis of aggressive B-cell lymphoma patients. Haematologica 2019;104:e25-8. [Crossref] [PubMed]
- Swiderska-Chadaj Z, Hebeda K, van den Brand M, et al. Predicting MYC translocation in HE specimens of diffuse large B-cell lymphoma through deep learning. Medical Imaging 2020: Digital Pathology. 2020. Available online:
10.1117/12.2549650 10.1117/12.2549650 - Swiderska-Chadaj Z, Hebeda KM, van den Brand M, et al. Artificial intelligence to detect MYC translocation in slides of diffuse large B-cell lymphoma. Virchows Arch 2021;479:617-21. [Crossref] [PubMed]
- Kluk MJ, Ho C, Yu H, et al. MYC Immunohistochemistry to Identify MYC-Driven B-Cell Lymphomas in Clinical Practice. Am J Clin Pathol 2016;145:166-79. [Crossref] [PubMed]
- Sukov WR, Hodge JC, Lohse CM, et al. TFE3 rearrangements in adult renal cell carcinoma: clinical and pathologic features with outcome in a large series of consecutively treated patients. Am J Surg Pathol 2012;36:663-70. [Crossref] [PubMed]
- Magers MJ, Udager AM, Mehra R. MiT Family Translocation-Associated Renal Cell Carcinoma: A Contemporary Update With Emphasis on Morphologic, Immunophenotypic, and Molecular Mimics. Arch Pathol Lab Med 2015;139:1224-33. [Crossref] [PubMed]
- Cheng J, Han Z, Mehra R, et al. Computational analysis of pathological images enables a better diagnosis of TFE3 Xp11.2 translocation renal cell carcinoma. Nat Commun 2020;11:1778. [Crossref] [PubMed]
- Owens MA, Horten BC, Da Silva MM. HER2 amplification ratios by fluorescence in situ hybridization and correlation with immunohistochemistry in a cohort of 6556 breast cancer tissues. Clin Breast Cancer 2004;5:63-9. [Crossref] [PubMed]
- Wolff AC, Hammond ME, Hicks DG, et al. Recommendations for human epidermal growth factor receptor 2 testing in breast cancer: American Society of Clinical Oncology/College of American Pathologists clinical practice guideline update. J Clin Oncol 2013;31:3997-4013. [Crossref] [PubMed]
- van der Logt EM, Kuperus DA, van Setten JW, et al. Fully automated fluorescent in situ hybridization (FISH) staining and digital analysis of HER2 in breast cancer: a validation study. PLoS One 2015;10:e0123201. [Crossref] [PubMed]
- Chea V, Pleiner V, Schweizer V, et al. Optimized workflow for digitalized FISH analysis in pathology. Diagn Pathol 2021;16:42. [Crossref] [PubMed]
- Lee B, Lee T, Lee SH, et al. Clinicopathologic characteristics of EGFR, KRAS, and ALK alterations in 6,595 lung cancers. Oncotarget 2016;7:23874-84. [Crossref] [PubMed]
- Warth A, Penzel R, Lindenmaier H, et al. EGFR, KRAS, BRAF and ALK gene alterations in lung adenocarcinomas: patient outcome, interplay with morphology and immunophenotype. Eur Respir J 2014;43:872-83. [Crossref] [PubMed]
- Saric M, Russo M, Stella M, et al. CNN-based Method for Lung Cancer Detection in Whole Slide Histopathology Images. The 4th International Conference on Smart and Sustainable Technologies (SpliTech), 2019. Available online:
10.23919/SpliTech.2019.8783041 10.23919/SpliTech.2019.8783041 - Wei JW, Tafe LJ, Linnik YA, et al. Pathologist-level classification of histologic patterns on resected lung adenocarcinoma slides with deep neural networks. Sci Rep 2019;9:3358. [Crossref] [PubMed]
- Yu KH, Zhang C, Berry GJ, et al. Predicting non-small cell lung cancer prognosis by fully automated microscopic pathology image features. Nat Commun 2016;7:12474. [Crossref] [PubMed]
- Pham HHN, Futakuchi M, Bychkov A, et al. Detection of Lung Cancer Lymph Node Metastases from Whole-Slide Histopathologic Images Using a Two-Step Deep Learning Approach. Am J Pathol 2019;189:2428-39. [Crossref] [PubMed]
- Coudray N, Ocampo PS, Sakellaropoulos T, et al. Classification and mutation prediction from non-small cell lung cancer histopathology images using deep learning. Nat Med 2018;24:1559-67. [Crossref] [PubMed]
- Conde E, Suárez-Gauthier A, Benito A, et al. Accurate identification of ALK positive lung carcinoma patients: novel FDA-cleared automated fluorescence in situ hybridization scanning system and ultrasensitive immunohistochemistry. PLoS One 2014;9:e107200. [Crossref] [PubMed]
- Hernandez S, Conde E, Prieto M, et al. P1.02-043 multiplexed fish (ALK/ROS1, Ret, NTRK1) in lung adenocarcinomas: Novel dual ALK/ros1 probe and automated scanning system. J Thorac Oncol 2017;12:S513-4. [Crossref]
- Höhne J, de Zoete J, Schmitz AA, et al. Detecting genetic alterations in BRAF and NTRK as oncogenic drivers in digital pathology images: towards model generalization within and across multiple thyroid cohorts. Proceedings of Machine Learning Research 2021;156. Available online: https://proceedings.mlr.press/v156/hohne21a/hohne21a.pdf
- Le NQK, Hung TNK, Do DT, et al. Radiomics-based machine learning model for efficiently classifying transcriptome subtypes in glioblastoma patients from MRI. Comput Biol Med 2021;132:104320. [Crossref] [PubMed]
- Le NQK, Kha QH, Nguyen VH, et al. Machine Learning-Based Radiomics Signatures for EGFR and KRAS Mutations Prediction in Non-Small-Cell Lung Cancer. Int J Mol Sci 2021;22:9254. [Crossref] [PubMed]
- Song Z, Liu T, Shi L, et al. The deep learning model combining CT image and clinicopathological information for predicting ALK fusion status and response to ALK-TKI therapy in non-small cell lung cancer patients. Eur J Nucl Med Mol Imaging 2021;48:361-71. [Crossref] [PubMed]
- Chang C, Sun X, Wang G, et al. A Machine Learning Model Based on PET/CT Radiomics and Clinical Characteristics Predicts ALK Rearrangement Status in Lung Adenocarcinoma. Front Oncol 2021;11:603882. [Crossref] [PubMed]
- Janowczyk A, Basavanhally A, Madabhushi A. Stain Normalization using Sparse AutoEncoders (StaNoSA): Application to digital pathology. Comput Med Imaging Graph 2017;57:50-61. [Crossref] [PubMed]
- Raciti P, Sue J, Ceballos R, et al. Novel artificial intelligence system increases the detection of prostate cancer in whole slide images of core needle biopsies. Mod Pathol 2020;33:2058-66. [Crossref] [PubMed]
- Bejnordi BE, Zuidhof G, Balkenhol M, et al. Context-aware stacked convolutional neural networks for classification of breast carcinomas in whole-slide histopathology images. J Med Imaging (Bellingham) 2017;4:044504. [Crossref] [PubMed]
- Chen CL, Chen CC, Yu WH, et al. An annotation-free whole-slide training approach to pathological classification of lung cancer types using deep learning. Nat Commun 2021;12:1193. [Crossref] [PubMed]
- Fraggetta F. Clinical-grade Computational Pathology: Alea Iacta Est. J Pathol Inform 2019;10:38. [Crossref] [PubMed]
宋俊权
复旦大学附属肿瘤医院(更新时间:2023-05-05)
(本译文仅供学术交流,实际内容请以英文原文为准。)
Cite this article as: Beretta C, Ceola S, Pagni F, L’Imperio V. The role of digital and integrative pathology for the detection of translocations: a narrative review. Precis Cancer Med 2022;5:16.