精准医学与肿瘤治疗的未来
精确医学(precision medicine)的概念可以追溯到20世纪60年代,Evans和Clark首次提出药物遗传学/药物基因组学(pharmacogenetics/pharmacogenomics)的概念[1]。随着二代测序(next generation sequencing, NGS)技术的发展,出现大量高通量数据,这一概念发展为“系统医学(systems medicine)”或“系统生物医学(systems biomedicine)”,并逐渐向“精确医学(precision medicine)”和“个体化医学(personalized medicine)”转变[1-5]。随着越来越多的基因组级数据分析和系统生物学数据的爆发,诸如“基因组时代医学(genomic-era medicine)”[6]、“预测性、预防性、个性化和参与性(P4)医学(Predictive, preventative, personalized and participatory, (P4)medicine)”[7]、“自我医学(Me medicine)”[8]、“P4系统医学(P4 systems medicine)”[9]和“计算系统生物医学(computational systems biomedicine)”[10]等术语也被赋予了相同的概念。从本质上讲,这涉及包含任何针对个体基因及其对生活方式和环境影响的相关治疗措施[11]。尽管“个体化医学”和“精准医学”在概念上几乎相同,但两者仍存在不同之处[12]。引用Ginsburg和Phillips的话,前者是一种考虑患者基因结构的同时,也关注患者的喜好、信念、态度、认知及社交等内容的治疗手段。相比之下,后者“描述了一个更加依赖于数据、分析和信息的医疗保健服务模式”[12]。然而,精准医学的概念,主要围绕快速识别新的药物靶点,以及解读导致或促进疾病遗传变异的特异性机制[13]。这一概念彻底改变了许多疾病的治疗模式。其中,值得一提的就是肿瘤,由于其存在异质性,早期开发的药物疗效不佳,肿瘤会持续生长[13]。由于基因的多效性,在强调识别新的抗肿瘤药物靶点的迫切需要的同时,也应考虑药物引发的不良反应[14]。
虽然研究在不断地发现新的抗肿瘤治疗蛋白靶点,但其治疗意义仍需要进一步探索。此外,高通量基因组学和蛋白质组学的发展,极大地帮助我们更好地认识涉及基因与环境之间复杂相互作用的癌症发病机制。基因之间的复杂相互作用可以产生多效性,即单个基因可影响同一机体的多个表型特征。因此,如果使用不同的药物不加选择地作用于某些基因,这些基因的相关蛋白产物也会受到影响。在这种情况下,人们尝试使用一套系统的方法去识别引起不良反应的新的肿瘤相关基因及蛋白[14]。但是,对于治疗像肿瘤这样的复杂疾病,更重要的是识别和治疗其根本病因。因此需要在基因组水平上进行详细的探索,以识别和验证一般肿瘤患者或肿瘤患者某个亚组的特定突变和/或其他基因组/转录组学改变。本质上,在不同类型的肿瘤中已观察到高频率的染色体畸变。这些畸变包括染色体易位、扩增和缺失、突变以及端粒延长[15]。在DNA序列和染色体水平上的大规模基因组变化,不仅肿瘤患者诊断时可见,而且会随着时间的推移不断演变。这表明了基因组的不稳定,也是大多数肿瘤的基本特征,与恶性表型的发生发展相关[15]。在上述肿瘤畸变中,目前已有对重组酶(RAD51)水平和同源重组活性升高的详细研究[15-17]。由DNA双链断裂(DNA double strand break, DSB)修复可激发基因组不稳定性。已证实,前列腺癌的基因组不稳定是由SPOP(斑点型POZ蛋白)突变引起的[18]。在尿路上皮癌中,对诸如L1细胞黏附分子(L1 cell adhesion molecule, L1CAM)和APOBEC等的蛋白克隆突变也有进一步的研究[19]。此类基因突变的增加和/或表达变化也可能促进肿瘤多药耐药性的产生,从而对精准癌症药物的开发造成潜在的阻碍。
过去数十年,由于数据共享的新倡议,大量临床和基础生物学研究数据可供使用,包括从基因组学、转录组学、蛋白质组学到代谢组学和相互作用组学的数据。因此,为了有效管理这一海量信息,一个新的研究领域逐渐出现,以提供国际化观点[20,21]。该研究领域被称为“系统生物学”,有助于理解疾病本身,实际上,疾病本身是众多分子受不同网络调节、不同途径影响的综合结果[21,22]。因此,疾病的系统生物学方法被认为是在疾病的系统水平上提供的鸟瞰视图。本质上,这将有助于识别不同的诊断生物标志物,并通过药物的发现、优化和组合开发干预治疗手段[20,21]。表1归纳了经不同生物技术处理、并保存在不同类型数据库中的大量实验数据。

Full table
为开发乳腺癌系统模型,Carels等人使用了不同组织学亚型的细胞系来了解激素受体的状态[23],包括雌激素受体(estrogen receptor,ER)、孕激素受体(progesterone receptor,PR)和人类表皮生长因子受体2(human epidermal growth factor receptor, HER2/ERBB2),这些分子可以为患者提供特异性治疗指征[23]。他们利用个性化治疗后的生存数据,绘制了Kaplan-Meier曲线图,阐明患者的5年生存率[23]。通过深入研究乳腺癌扩散过程中的分子改变,他们提出了一个改良版本的肿瘤异质性进化过程,包括基因组不稳定性和克隆扩展[23]。有研究尝试进一步利用患者的肿瘤样本帮助确定有效药物和/或其组合,用于进一步的临床试验评估和个性化治疗[11]。为此,Pauli等人整合了全外显子序列与56个肿瘤来源的类器官以及19例子宫恶性肿瘤和结肠癌患者源性的异种移植(PDX)模型[11]。此外,整合传统天然药物和合成西药可获取生物学数据,已被认为是肿瘤精准医学研究的有效替代策略。由于不同表型和基因的细胞亚群,使肿瘤产生异质性,这一策略更具有其必要性[24]。Efferth等人利用网络药理学方法,分析了新香豆素或海香豆中提取的大明香对白血病细胞三种不同基因靶点的影响。它们是所有天然产物、治疗性抗体和合成小分子的主要成分、反应物和效应物[24]。他们认为,这种整合主要原因在于天然药物是作为一系列具有多种特异性物质的混合物,可以同时作用于不同的靶点[24]。这些天然药物是复方药的本质,主要用于老年患者[25,26]。然而,正如Finkelstein等人报告的那样,同时使用17~26种药物可能存在与多种药物副作用相关的风险[27]。尽管是在完全不同的背景下进行的研究,患者同时患有慢性心肺部疾病,但这表明了复杂的药物与药物、药物与基因之间的相互作用,从而说明了多药联合的风险[26]。此外,多药联合的情况下,需要精确识别几种小分子药物的不良反应。多种小分子药物联用引起的非预期副作用,通常被称为药物不良反应(adverse drug reactions, ADRs),其作用机制及其在不同人群的反应严重程度各不相同[27]。对此类不良反应的评估在很大程度上取决于来自规模有限的临床试验和存在偏倚的上市后报告,这些报告倾向于低估患者风险[27]。为了克服药物不良反应低估相关的风险,研究者们应用了系统药理学。这需要一个详细的网络分析,涉及整合化学和生化实体(如饮食中的药物和食物分子)与包含基因和蛋白质的生物实体之间的相互作用。Boland等人使用二分图阐明了基于系统药理学各个成分之间的相关联系[27]。
随着时间的推移,肿瘤的治疗已发生了一些变化以精准而有效地满足大众的需要,包括识别特定基因突变,引起起源部位不可控增殖的特定基因功能的存在或缺失和/或染色体重排。然而,尽管做出了这些努力,但肿瘤内异质性和持续存在的基因组进化使得筛选试别可能受益于特定肿瘤治疗的患者变得复杂[28]。在维持染色体结构和功能方面起关键作用的表观遗传修饰也开始成为新兴技术关注的焦点[28]。这些策略尝试整合受基因和表观遗传修饰的相互作用调节的恶性转化的概念[28]。恶性转化的基因和表观遗传修饰机制的识别加快了发现导致疾病的遗传变异,促进针对这些变异的药物开发进程。然而,除了多态性(次要等位基因频率,minor allele frequency,MAF>1%)外,大量遗传变异,包括罕见突变(MAF<1%),亦参与调节对药物反应的真正个性化预测[29]。除了肿瘤基因组中的胚系多态性作为药物遗传学信息指示外,此类体细胞变体已开始用于某些药物/基因对[29]。SuarezKurtz从一项巴西的前瞻性研究[29]中,回顾并证实了临床药物遗传学实施联盟(Clinical Pharmacogenetics Implementation Consortium))和/或荷兰药物遗传学工作组(Dutch Pharmacogenetic Working Group)指导的伊立替康/UGT1A1、三苯氧胺/CYP2D6、硫嘌呤/TPMT和氟嘧啶/UGT1A1等药物/基因对的临床价值[30]。
因此,上述讨论让我们认识到,将数据科学和分析整合到药物遗传学和/或药物基因组学,是为精确癌症医学铺平道路的必要条件。反过来,这将通过优化工具和信息开发,为改善肿瘤患者预后提供帮助。这甚至可以在当地和全球范围内制定新的经济政策和议程,以促进这种新的医疗模式被采用[8]。
人类遗传学和药物反应之间的直接联系,就其共变异而言,已被整合到最大的人工管理数据库中,即药物基因组学知识库(Pharmacogenomics Knowledge Base, PharmGKB)[31]。然而,由于生物医学术语和知识的浩瀚、动态性和复杂性,提取这些已发表的数据,并转化为有意义和可翻译的信息,是一项艰巨的任务[32]。因此,为了实现最早的精准医学,除了统计学之外,还采用了依靠机器学习的方法,将基因与药物联系起来[33]。事实上,Chang和Altman[34]已使用统计方法确定共现,使用典型的机器学习方法,即自然语言处理(Natural Language Processing, NLP),对从文献中提取的药物基因对进行分类。随后,Garten和Altman通过一个名为Pharmspresso的文本挖掘工具提取了Phramacogenomics (PGx)的特定概念之间的关系[35]。此外,Theobald等人根据早期报告的共现统计数据,创建了条件概率表,将其转换为n-way贝叶斯网络,以分析生物实体之间的关系[36]。尽管如此,NLP系统的开发仍在实践中,通过Enhanced SemRep, Syntactic Dependency Structure和其他方法,从MEDLINE引文和摘要的共现中揭示药物-基因关系[37-39]。
随着对精确肿瘤医学的关注,人们还努力按照潜在的药物特异性对PGx重要基因进行排序[40,41]。事实上,目前已报道了一种半监督迭代搜索和排序(bootstrapping)算法,其目的是对PGx基因进行优先级排序[32,42]。该算法使用2000万条MEDLINE摘要和PGx特异基因,经过两次迭代后,精度达0.219,对于排名靠前的配对,最终精度增加至0.561。在精确度、召回率或F1(精确度:0.251 vs 0.152,召回率:0.396 vs 0.856和F1:0.292 vs 0.254)方面,与非PGx特异性种子或MEDLINE共现术语相比,获得较大提升[32,42]。在此,我们回顾一下精确度、召回率和F1这几个术语。精确度是指真阳性与总阳性(包括假阳性)的比率,召回率是指真阳性与真实阳性(包括假阴性)的比率,F1是指准确率与召回率的加权平均。当预测的药物基因对与药物不良事件进行比较时,可获得很强的相关性[42]。
在研究药物-基因关系,按照它们在治疗中的相关性对其进行排序的同时,人们也在努力绘制癌细胞进化图谱,通常被称为克隆进化,包括肿瘤DNA纯度和癌细胞倍数性[43]。然而,由于可能由进展或治疗而导致的肿瘤异质性,全局优化的方法[44-47]可能无法评估潜在细胞群的复杂性,因为这些方法忽略了基因组多样性[43]。因此,综合考虑有利于确定有体细胞缺失的SNP的等位基因频率值的三种类型细胞群,即非肿瘤细胞群、有缺失的肿瘤细胞群和无确实的细胞群,有助于研究人员开发一种局部优化方法[43]。Prandi等人利用观察到的AFs的概率分布开发的模型,包含了21例肺腺癌[48]、24例转移性黑色素瘤[49]和55例原发性前列腺癌[50]的肿瘤样本的全基因组测序(WGS)数据,被称为CLONET(CLONality Estimate in Tumors, 肿瘤中的克隆性估计),可以计算整个基因组中所有缺失的局部混合值,揭示特定的病变。
“组学”数据驱动分析、系统生物学和数学建模方法的综合效应已在改变治疗模式。该模型被称为OnTrack,除了维持肿瘤细胞3D培养和小鼠异种移植模型外,旨在记录原发肿瘤、转移瘤和配对的健康人组织样本对药物的反应[51]。这有助于对结肠癌患者的全基因组、外显子组、转录组、甲基化组和全蛋白组学的相应组学数据进行治疗前和治疗后分析[51]。整体协同效应有助于预测患者的反应,及时预测耐药性[51]。类似的基因组数据的大规模整合,例如IGNITE,已经开始用于构建多个电子健康记录(Electronic Health Record)网络[52,53]。美国国立卫生研究(NIH)院资助成立的IGNITE(Implementing GeNomics In practice, 在实践中实施基因组学;www.IGNITE-GeNomics.org)网络成立于2013年,拥有一个协作中心,负责管理六个项目,用于开发、调查和传播基因组医学实践模型,并提供临床决策支持(clinical decision support, CDS)。IGNITE网络的一些中心(如Vanderbilt University, 范德比尔特大学)保留了CDS的外部链接(如MyCancerGenome.org)[52]。
此外,针对癌症的大规模生物医学数据整合已在实践中得到应用,以分析和提供多方面的指导学习和决策的用户平台。其中一个案例就是G-DOC Plus,该项目整合了来自不同公共数据库和私有资源的超过10000个患者的数据,包括Gene Expression Omnibus (GEO)、The Cancer Genome Atlas (TCGA)、Repository for Molecular BRAin Neoplasia DaTa (REMBRANDT)、肺癌和结肠癌的caArray研究、ImmPort以及1000个基因组数据集[54]。G-DOC Plus工具已被广泛用于癌症和非癌症研究,通过多组学分析研究体细胞突变和肿瘤MRI图像,挖掘生物标记物,提供生物信息学、计算和科学相关领域的知识培训[54]。此外,通过IDICAP提供的网络工具,可被用于整合基于肿瘤小组的综合药物干预[55]。从本质上来说,除了整合与试验药物及其位置、具有已知基因靶点的药物、任何已知单核苷酸多态性(SNP)效应和其他疾病条件相关的事件和数据外,这些工具还把乳腺癌、卵巢癌和常见肿瘤的基因信息与ClinicalTrials.gov以及DrugBank网站上的药物和临床试验数据进行了整合[55,56]。与药物-基因相互作用数据库(Drug Gene Interaction Database ,DGIdb),甚至ClinicalTrials.gov的类似结果相比,IDICAP被认为具有足够精确性,可以指导临床试验和药物[55,57]。所有这些大型数据整合工具都使精准肿瘤医学向前迈出了一步。
将精准肿瘤医学从多分支分析方法转变为基于网络分析的简化案例,并与相关生物信息学方法相结合,有助于研究人员确定多靶点药物疗法,并在三阴性乳腺癌(TNBC)模型和其他模型中对其进行优先级区分[58-61]。Vitali等人从某些肿瘤类型/亚型开始,有效地从理论上分析了疾病特异性蛋白质相互作用(Protein-Protein Interaction,PPI)网络,该网络通过整合不同的数据库和知识库来确定一系列潜在感兴趣的药物靶点组合[58]。应用一种基于三因子分解矩阵的方法,结合现有关于所选药物作用机制的认知,在TNBC中通过多项体外实验进行验证[58]。另一项关于TNBC模型的研究是一个名为GenEx-TNBC的计算机平台,该平台通过基于网络的方法,对数千种经批准的和实验性的药物对TNBC每个分子亚型的治疗潜力进行优先级排序[59]。从本质上讲,网络是基于患者和细胞系的基因表达数据构建的,并与已知药物蛋白靶点的药物作用网络进行统计学相符性分析[59]。通过GenEx-TNBC的方法成功定义药物,并对其分类进行进一步体外试验验证,从而建立首个将药物与疾病相关联的平台[59]。应用此类已知的、预测蛋白-蛋白相互作用的数据库,如STRING[62],以及蛋白-药物相互作用的数据库,如STITCH[63],可以构建基于统计学检验的证据网络,以检测统计学差异,即差异相关性评估(Evaluation of Differential DependencY,EDDY)[60]。EDDY以公共门户EDDY-CTRP的形式呈现,其中包含了肿瘤治疗反应门户(Cancer Therapeutics Response Portal,CTRP,一个具有400多个小分子的药物反应测量的数据集)的数据以及肿瘤细胞系百科全书(Cancer Cell Line Encyclopedia,CCLE)中细胞系的RNAseq数据[60]。Tran等人认为,他们构建的证据网络可以发现约70%的药物-介质对的方法,有可能为药物敏感性提供重要的信息[60]。事实上,利用几个这样的蛋白质、药物和/或基因的分子网络,已帮助研究人员在过去十年中通过网络拓扑分析和其他图论手段确定潜在的治疗靶点和通路[61]。Ashraf等人最近报告的一项工作,使用了不同的中心性度量进行比较和对比,并确定了没有潜在副作用的靶点[14]。本质上,他们已经证明了特征向量中心性(C)以及其他网络参数,如k-core(k)和功能模块性(functional modularity,F)的有效性,从而提出了KFC标准,作为确定药品库(Drugbank)中反映的上市药物副作用的关键[14,56]。表2记录了从不同肿瘤类型中发现的几种基因-药物相互作用。

Full table
当世界各地的研究人员非常积极地使用一些统计和数学参数和方法来分析巨量的生物数据,以发展精准肿瘤医学时,生物信息学程序员在满足他们的需求。由于基因组变异,以及伴随的临床和表型特征,可以通过NGS得到有效识别,潜在的工作流程和方法开始涌入,以识别潜在的基因靶点及其相应的药物[64]。此外,R/Bioconductor包rDGIdb使R用户能够拥有现成的包、资源和工具,用于与具有来自15种不同资源的药物-基因相互作用的DGIdb一起处理此类NGS数据[57,64]。此外,另一个R包,即肿瘤变异探索者(Cancer Variant Explorer,CVE),利用Oncotator和DGIdb,提供单个或多个肿瘤外显子组的变异信息,以便识别变异人群中的驱动突变及其耐药机制,从本质上评估靶点的可能潜在药物[65,66]。根据Mock等人的研究,CVE使研究人员能够对组织特异性网络进行探索性分析,以进行转化研究[65]。最后,在一项正在进行的预测药物及其靶点的尝试中,采用了一种以成对方式预测药物的新方法。该方法是一种基于核(Kernel-based)的机器学习方法和多核学习(multiple kernel learning,MKL)方法,被称为成对MKL[67]。Cichonska等人利用了多达167995个生物活性测量值和3120个成对核,基于该方法评估了准确预测药物在一系列肿瘤细胞系中的抗肿瘤作用以及抗肿瘤药物在其kinome靶空间中的靶点分布的表现[67]。
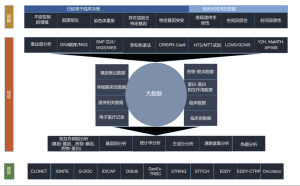
总之,目前的新技术可以评估整个基因组的遗传、表观遗传和转录组学变化(图1)。此外,随着生物信息学的进步,我们现在可以整合不同类型的数据以确定肿瘤基因组变化以及潜在的突变过程(图1)。这为在特定肿瘤或肿瘤亚组人群中,鉴别特定肿瘤的诊断/预后标志物以及治疗靶点奠定了基础(图1)。在患者个体层面应用这些技术可能有助于为个体选择正确的药物、药物组合和/或治疗策略。尽管仍需要消除/最小化与这些技术相关的一些限制,但它们为个性化和精确医疗提供了基础。以网络生物学为基础的预测将引领全球进入精确肿瘤医学的全新时代,精确肿瘤医学可以满足抗击这种致命疾病的个人需要。
Acknowledgments
The authors acknowledge the support of Sunway University, Selangor, Malaysia for providing the computational facilities.
Funding: None.
Footnote
Provenance and Peer Review: This article was commissioned by the Guest Editor Masood A. Shammas for the series “Genomic Instability, Clonal Evolution and Oncogenesis” published in Precision Cancer Medicine. The article has undergone external peer review.
Conflicts of Interest: All authors have completed the ICMJE uniform disclosure form (available at http://dx.doi.org/10.21037/pcm.2019.09.01). The series “Genomic Instability, Clonal Evolution and Oncogenesis” was commissioned by the editorial office without any funding or sponsorship. The authors have no other conflicts of interest to declare.
Ethical Statement: The authors are accountable for all aspects of the work in ensuring that questions related to the accuracy or integrity of any part of the work are appropriately investigated and resolved.
Open Access Statement: This is an Open Access article distributed in accordance with the Creative Commons Attribution-NonCommercial-NoDerivs 4.0 International License (CC BY-NC-ND 4.0), which permits the non-commercial replication and distribution of the article with the strict proviso that no changes or edits are made and the original work is properly cited (including links to both the formal publication through the relevant DOI and the license). See: https://creativecommons.org/licenses/by-nc-nd/4.0/.
References
- Duffy DJ. Problems, challenges and promises: perspectives on precision medicine. Brief Bioinform 2016;17:494-504. [Crossref] [PubMed]
- Zhang Z, Zhao Z, Liu B, et al. Systems biomedicine: it’s your turn-recent progress in systems biomedicine. Quant Biol 2013;1:140-55. [Crossref]
- Kamada T. System biomedicine: a new paradigm in biomedical engineering. Front Med Biol Eng 1992;4:1-2. [PubMed]
- Wasi P. Human genomics: implications for health. Southeast Asian J Trop Med Public Health 1997;28:19-24. [PubMed]
- Langreth R, Waldholz M. New era of personalized medicine targeting drugs for each unique genetic profile. Oncologist 1999;4:426-7. [PubMed]
- Ashburn TT, Wilson SK, Eisenstein BI. Human tissue research in the genomic era of medicine: balancing individual and societal interests. Arch Intern Med 2000;160:3377-84. [Crossref] [PubMed]
- Hood L, Heath JR, Phelps ME, et al. Systems biology and new technologies enable predictive and preventative medicine. Science 2004;306:640-3. [Crossref] [PubMed]
- Dickenson D. Me medicine vs. we medicine: Reclaiming biotechnology for the common good. Columbia University Press, 2013.
- Cesario A, Auffray C, Russo P, et al. P4 medicine needs P4 education. Curr Pharm Des 2014;20:6071-2. [Crossref] [PubMed]
- Dubitzky W. Understanding the computational methodologies of systems biology. Brief Bioinform 2006;7:315-7. [Crossref] [PubMed]
- Pauli C, Hopkins BD, Prandi D, et al. Personalized in vitro and in vivo cancer models to guide precision medicine. Cancer Discov 2017;7:462-77. [Crossref] [PubMed]
- Ginsburg GS, Phillips KA. Precision medicine: From science to value. Health Aff (Millwood) 2018;37:694-701. [Crossref] [PubMed]
- Dugger SA, Platt A, Goldstein DB. Drug development in the era of precision medicine. Nat Rev Drug Discov 2018;17:183-96. [Crossref] [PubMed]
- Ashraf MI, Ong SK, Mujawar S, et al. A side-effect free method for identifying cancer drug targets. Sci Rep 2018;8:6669. [Crossref] [PubMed]
- Shammas MA, Shmookler Reis RJ, Koley H, et al. Dysfunctional homologous recombination mediates genomic instability and progression in myeloma. Blood 2009;113:2290-7. [Crossref] [PubMed]
- Pal J, Bertheau R, Buon L, et al. Genomic evolution in Barrett's adenocarcinoma cells: critical roles of elevated hsRAD51, homologous recombination and Alu sequences in the genome. Oncogene 2011;30:3585-98. [Crossref] [PubMed]
- Lu R, Pal J, Buon L, et al. Targeting homologous recombination and telomerase in Barrett’s adenocarcinoma: impact on telomere maintenance, genomic instability and tumor growth. Oncogene 2014;33:1495-505. [Crossref] [PubMed]
- Boysen G, Barbieri CE, Prandi D, et al. SPOP mutation leads to genomic instability in prostate cancer. Elife 2015;4:e09207. [Crossref] [PubMed]
- Faltas BM, Prandi D, Tagawa ST, et al. Clonal evolution of chemotherapy-resistant urothelial carcinoma. Nat Genet 2016;48:1490-99. [Crossref] [PubMed]
- Chen B, Butte AJ. Leveraging big data to transform target selection and drug discovery. Clin Pharmacol Ther 2016;99:285-97. [Crossref] [PubMed]
- Wang RS, Maron BA, Loscalzo J. Systems medicine: evolution of systems biology from bench to bedside. Wiley Interdiscip Rev Syst Biol Med 2015;7:141-61. [Crossref] [PubMed]
- Lahiri C. Disease complexity-A bird’s eye view. J Infect Dis Ther 2015;3:53.
- Carels N, Spinassé LB, Tilli TM, et al. Toward precision medicine of breast cancer. Theor Biol Med Model 2016;13:7. [Crossref] [PubMed]
- Efferth T, Saeed ME, Mirghani E, et al. Integration of phytochemicals and phytotherapy into cancer precision medicine. Oncotarget 2017;8:50284-304. [Crossref] [PubMed]
- Hao DC, Xiao PG. Network pharmacology: a Rosetta Stone for Traditional Chinese Medicine 2014;75:299-312.
- Finkelstein J, Friedman C, Hripcsak G, et al. Potential utility of precision medicine for older adults with polypharmacy: a case series study. Pharmgenomics Pers Med 2016;9:31-45. [Crossref] [PubMed]
- Boland MR, Jacunski A, Lorberbaum T, et al. Systems biology approaches for identifying adverse drug reactions and elucidating their underlying biological mechanisms. Wiley Interdiscip Rev Syst Biol Med 2016;8:104-22. [Crossref] [PubMed]
- Coyle KM, Boudreau JE, Marcato P. Genetic mutations and epigenetic modifications: driving cancer and informing precision medicine. Biomed Res Int 2017;2017:9620870. [Crossref] [PubMed]
- Suarez-Kurtz G. Pharmacogenetic testing in oncology: a Brazilian perspective. Clinics (Sao Paulo) 2018;73:e565s. [Crossref] [PubMed]
- Relling MV, Klein TE. CPIC: clinical pharmacogenetics implementation consortium of the pharmacogenomics research network. Clin Pharmacol Ther 2011;89:464-7. [Crossref] [PubMed]
- Klein TE, Chang JT, Cho MK, et al. Integrating genotype and phenotype information: an overview of the PharmGKB project. Pharmacogenomics J 2001;1:167-70. [Crossref] [PubMed]
- Xu R, Wang Q. An iterative searching and ranking algorithm for prioritising pharmacogenomics genes. Int J Comput Biol Drug Des 2013;6:18-31. [Crossref] [PubMed]
- Garten Y, Coulet A, Altman RB. Recent progress in automatically extracting information from the pharmacogenomic literature. Pharmacogenomics 2010;11:1467-89. [Crossref] [PubMed]
- Chang JT, Altman RB. Extracting and characterizing gene-drug relationships from the literature. Pharmacogenetics 2004;14:577-86. [Crossref] [PubMed]
- Garten Y, Altman RB. Pharmspresso: a text mining tool for extraction of pharmacogenomic concepts and relationships from full text. BMC bioinformatics 2009;10:S6. [Crossref] [PubMed]
- Theobald M, Shah N, Shrager J. Extraction of conditional probabilities of the relationships between drugs, diseases, and genes from PubMed guided by relationships in PharmGKB. Summit Transl Bioinform 2009;2009:124-8. [PubMed]
- Ahlers CB, Fiszman M, Demner-Fushman D, et al. Extracting semantic predications from Medline citations for pharmacogenomics. Pac Symp Biocomput 2007;2007:209-20. [PubMed]
- Coulet A, Shah NH, Garten Y, et al. Using text to build semantic networks for pharmacogenomics. J Biomed Inform 2010;43:1009-19. [Crossref] [PubMed]
- Xu R, Wang Q. A knowledge-driven conditional approach to extract pharmacogenomics specific drug–gene relationships from free text. J Biomed Inform 2012;45:827-34. [Crossref] [PubMed]
- Hansen NT, Brunak S, Altman RB. Generating genome‐scale candidate gene lists for pharmacogenomics. Clin Pharmacol Ther 2009;86:183-9. [Crossref] [PubMed]
- Wu Y, Liu M, Zheng WJ, et al. Ranking gene-drug relationships in biomedical literature using latent dirichlet allocation. Pac Symp Biocomput 2012;2012:422-33. [PubMed]
- Xu R, Wang Q. A semi-supervised approach to extract pharmacogenomics-specific drug–gene pairs from biomedical literature for personalized medicine. J Biomed Inform 2013;46:585-93. [Crossref] [PubMed]
- Prandi D, Baca SC, Romanel A, et al. Unraveling the clonal hierarchy of somatic genomic aberrations. Genome Biol 2014;15:439. [Crossref] [PubMed]
- Van Loo P, Nordgard SH, Lingjærde OC, et al. Allele-specific copy number analysis of tumors. Proc Natl Acad Sci U S A 2010;107:16910-5. [Crossref] [PubMed]
- Greenman CD, Bignell G, Butler A, et al. PICNIC: an algorithm to predict absolute allelic copy number variation with microarray cancer data. Biostatistics 2010;11:164-75. [Crossref] [PubMed]
- Yau C, Mouradov D, Jorissen RN, et al. A statistical approach for detecting genomic aberrations in heterogeneous tumor samples from single nucleotide polymorphism genotyping data. Genome Biol 2010;11:R92. [Crossref] [PubMed]
- Li A, Liu Z, Lezon-Geyda K, et al. GPHMM: an integrated hidden Markov model for identification of copy number alteration and loss of heterozygosity in complex tumor samples using whole genome SNP arrays. Nucleic Acids Res 2011;39:4928-41. [Crossref] [PubMed]
- Imielinski M, Berger AH, Hammerman PS, et al. Mapping the hallmarks of lung adenocarcinoma with massively parallel sequencing. Cell 2012;150:1107-20. [Crossref] [PubMed]
- Berger MF, Hodis E, Heffernan TP, et al. Melanoma genome sequencing reveals frequent PREX2 mutations. Nature 2012;485:502-6. [Crossref] [PubMed]
- Baca SC, Prandi D, Lawrence MS, et al. Punctuated evolution of prostate cancer genomes. Cell 2013;153:666-77. [Crossref] [PubMed]
- Henderson D, Ogilvie LA, Hoyle N, et al. Personalized medicine approaches for colon cancer driven by genomics and systems biology: OncoTrack. Biotechnol J 2014;9:1104-14. [Crossref] [PubMed]
- Weitzel KW, Alexander M, Bernhardt BA, et al. The IGNITE network: a model for genomic medicine implementation and research. BMC Med Genomics 2016;9:1. [Crossref] [PubMed]
- Simmons M, Singhal A, Lu Z. Text mining for precision medicine: bringing structure to EHRs and biomedical literature to understand genes and health. Adv Exp Med Biol 2016;939:139-66. [Crossref] [PubMed]
- Bhuvaneshwar K, Belouali A, Singh V, et al. G-DOC Plus - an integrative bioinformatics platform for precision medicine. BMC Bioinformatics 2016;17:193. [Crossref] [PubMed]
- Kosarek N, Ho ES. IDICAP: a novel tool for integrating drug intervention based on cancer panel. J Pers Med 2016;6:19. [Crossref] [PubMed]
- Law V, Knox C, Djoumbou Y, et al. DrugBank 4.0: shedding new light on drug metabolism. Nucleic Acids Res 2014;42:D1091-7. [Crossref] [PubMed]
- Cotto KC, Wagner AH, Feng YY, et al. DGIdb 3.0: a redesign and expansion of the drug–gene interaction database. Nucleic Acids Res 2018;46:D1068-73. [Crossref] [PubMed]
- Vitali F, Cohen LD, Demartini A, et al. A network-based data integration approach to support drug repurposing and multi-target therapies in triple negative breast cancer. PLoS One 2016;11:e0162407. [Crossref] [PubMed]
- Wathieu H, Issa NT, Fernandez AI, et al. Differential prioritization of therapies to subtypes of triple negative breast cancer using a systems medicine method. Oncotarget 2017;8:92926-42. [Crossref] [PubMed]
- Tran HJ, Speyer G, Kiefer J, et al. Contextualization of drug-mediator relations using evidence networks. BMC Bioinformatics 2017;18:252. [Crossref] [PubMed]
- Hao T, Wang Q, Zhao L, et al. Analyzing of molecular networks for human diseases and drug discovery. Curr Top Med Chem 2018;18:1007-14. [Crossref] [PubMed]
- Szklarczyk D, Franceschini A, Wyder S, et al. STRING v10: protein–protein interaction networks, integrated over the tree of life. Nucleic Acids Res 2015;43:D447-52. [Crossref] [PubMed]
- Kuhn M, Szklarczyk D, Franceschini A, et al. STITCH 3: zooming in on protein–chemical interactions. Nucleic Acids Res 2012;40:D876-80. [Crossref] [PubMed]
- Thurnherr T, Singer F, Stekhoven DJ, et al. Genomic variant annotation workflow for clinical applications. F1000Res 2016;5:1963. [Crossref] [PubMed]
- Mock A, Murphy S, Morris J, et al. CVE: an R package for interactive variant prioritisation in precision oncology. BMC Med Genomics 2017;10:37. [Crossref] [PubMed]
- Ramos AH, Lichtenstein L, Gupta M, et al. Oncotator: cancer variant annotation tool. Hum Mutat 2015;36:E2423-9. [Crossref] [PubMed]
- Cichonska A, Pahikkala T, Szedmak S, et al. Learning with multiple pairwise kernels for drug bioactivity prediction. Bioinformatics 2018;34:i509-18. [Crossref] [PubMed]

潘肖芬,中山大学附属第七医院
工作经历:
1.2020-1至现在, 中山大学,附属第七医院, 肿瘤科, 主治医师
2.2017-9至2019-12, 中山大学,附属第七医院, 肿瘤科, 医师
3.2015-7至2017-8,广东医科大学,附属医院,肿瘤中心,医师
科研成绩:
1.主持省课题1项,项目编号: 2020A1515110923
2.以第一作者或共同第一作者身份发表SCI论文6篇。
3.取得发明专利一项(发明人),专利号: ZL 2020 1 0091281.1(更新时间:2021/9/29)

薛冉
北京大学肿瘤医院。医学博士,北京大学肿瘤医院消化肿瘤内科/I期临床病区医师。长期从事肝胆胰疾病相关临床及基础研究工作。近年主持国家自然科学青年基金1项,北大医学青年培育基金A类1项,美捷登青年科学家基金1项。作为第一作者已发表SCI文章17篇,IF累计超过60分。现任《J Clin Transl Hepatol》及《中华生物医学工程》杂志青年编委,先后应邀成为Journal of Cellular and Molecular Medicine、Clinical Science、Bioscience Reports等杂志审稿人。受邀于APASL、DDW、ISVHLD Global Hepatitis Summit、UEG等国际会议进行英文口头汇报及壁报展示多次。(更新时间:2021/9/29)
(本译文仅供学术交流,实际内容请以英文原文为准。)
Cite this article as: Lahiri C, Pawar S, Mishra R. Precision medicine and future of cancer treatment. Precis Cancer Med 2019;2:33.